CS 1027 Computer Science Fundamentals II
Hello, dear friend, you can consult us at any time if you have any questions, add WeChat: daixieit
CS 1027
Computer Science Fundamentals II
Learning Outcomes
To gain experience with:
● Using Iterators
● Using stacks, queues, and iterators in coordination
● Creating and manipulating many objects to aid in solving problems
● Using an interface to handle similar tasks.
● Algorithm implementation
Introduction
DNA sequences are being used in many fields: genetics, virology, forensics, and even computing. Analysing the sequences is of critical importance. The sequences are composed of
four bases:adenine(A),thymine(T),guanine(G), andcytosine(C). A double helix of base pairs (A binds with T and G binds with C) form within a DNA molecule. In a DNA sequence, a triplet of base pairs form a codon which is used to code for amino acids as well as activation and termination. For this assignment, we are going to model DNA simply as a sequence of chars that we will traverse looking for certain patterns and pattern types. To limit the scope of our DNA analysis,
we are only going to focus on two pattern types: palindromes of a certain length and repeating sequences of a certain length and number of repetitions. In general, a palindrome is a sequence that reads the same forward as backwards. In English, well known palindromes are “race car” and “Madam, I'm Adam” (ignoring punctuation and spaces). In DNA, here are some examples: GGTACATGG, TAT, and ATGCCGTA. A notable feature of these palindromes are their lengths: 9, 3, and 8. The other pattern type we will focus on is sequences that repeat a certain number of times. For instance, we could look for a sequence of length 3 that repeats 4 times. Here are
some possible sequences that have that pattern: GGAGGAGGAGGA, TTTTTTTTTTTT, and CATCATCATCAT.
There will be a number of classes that will be used to build our DNA analyser tool: a DNA class which will provide us with arbitrary sequences which will simulate real DNA sequences, a palindrome checker that will recognize a palindrome of a certain length, a repeat checker that will recognize sequences of a certain lengths that are repeated a certain number of times, and an analyzer that takes a sequence check for specific types of sequences. The two checker classes will be used to create many instances, each will only check for sequences that start at a particular base in the sequence. Specifically, a new checker will be created for each base in the sequence. So if a DNA sequence of 100 bases long is to be analyzed for palindromes of length 7, palindromes of length 9, and a sequence of length 5 repeated twice, then the number of checkers created will be 100 times 3 (the number of pattern types searched for). This strategy of using many checkers helps reduce the complexity of code of each checker.
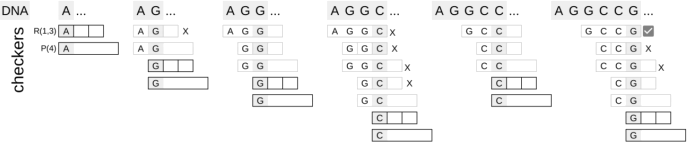
This picture shows a DNA sequence checked for two patterns: a single character repeated 3 times and a palindrome of length 4. The progression shows 6 different repeat checkers (these are the ones depicted by 3 squares): one for each new letter. Similarly, there are 6 different palindrome checkers (these are the ones depicted by longer rectangles). Each checker persists (the lighter bordered rectangles) until it fails or finds the pattern. So far in the DNA, there is only one found pattern: a palindrome of length 4 at the third location.
Provided files
The following is a list of files provided to you for this assignment. Do not alter the "Test" files since it may give you a false sense that your code is correctly performing.
● ArrayStack.java
● StackADT.java
● LinkedQueue.java
● QueueADT.java
● EmptyCollectionException.java
● LinearNode.java
● +
● DNA.java
● DnaTester.java
Classes to Implement
For this assignment, you must implement one interface and three Java classes: Checker, PalindromeChecker, RepeatChecker, DnaAnalyser. Follow the guidelines for each one below.
In all these classes, you can implement more private (helper) methods, if you want to, but you may not implement more public methods. You may not add instance variables other than the ones specified below nor change the variable types or accessibility (i.e. making a variable public when it should be private). Penalties will be applied if you implement additional instance variables or change the variable types or modifiers from what is described here.
DNA.java
This is a complete class but it contains a few methods that you will need to use. Its main purpose of this class is to produce a sequence of Characters one at a time to simulate a DNA sequence. It implements the iterator interface, hasNext() and next(), to have access to the sequence one Character at a time. As a means of testing defined sequences, the iterateString method produces an iterator that will walk through the input String. To help verify the results, the display method formats a long sequence into 50 character lengths with additional numbers to help indicate a characters’ location in the sequence.
● public static Iterator<Character> iterateString(String) - to simulate the DNAs action with defined String. The returned Iterator will give the characters of the input String one at a time.
● public static String display(String) - shows the DNA String in a friendlier way to locate the found patterns to better understand the results from the checkers
Checker.java
This is an interface that we use to define the important features of Checkers. The interface must have the following abstract methods:
● public boolean process(char c) - used to process a single char c
● public boolean finished() - used for seeing if the checker needs to continue processing
● public Checker cloneHere(int pos) - used to create a new Checker with the same attributes but restarting at pos position in the DNA sequence
PalindromeChecker.java
This class is used to check if a sequence of chars(passed to its process method) is a palindrome of a certain length
The class must have the following private instance variables:
● seqLength (final int) - holds the sought after length
● position (final int) - holds the start position in the DNA sequence (for tag reference)
● fail (boolean) - holds whether the processed sequence so far could not be palindrome
● patternSoFar (String) - holds the accumulated chars so far given to this checker
● previous (StackADT<Character>) - holds the initial chars that must later be checked
against the later chars to determine if the sequence is a palindrome
The class must have two constructors, implement the Checker interface, and override the toString method:
● public PalindromeChecker(int seqLength)
o Initializes all instance variables: seqLength as specified, position as 0, fail as false, patternSofar as “”, and previous as an empty stack
● private PalindromeChecker(int seqLength, int position)
o Same as above but position is specified. This constructor is only used in the cloneHere method.
● public boolean process(char c)
o If the palindrome has an even length
▪ For the first half of the characters, add them to the stack.
▪ For the second half of the characters, see if they match the expected characters on the stack, if not, set the instance variable, fail, to true.
o If the palindrome has an odd length do the same as the even except skip over the middle character (in terms of the stack)
o Return true if current length of the patternSoFar is the seqLength and fail is false
(meaning: it found a palindrome of the sought after length), otherwise return false.
● public boolean finished()
o Return fail
● public Checker cloneHere(int pos)
o Return a new PalindromeChecker with pos
● public String toString()
o Return a String representation of this checker (see DnaTest.java for details)
o e.g. “Palindrome(10) - 39{CAAGAAGAAC}”
RepeatChecker.java
This class is used to check if a sequence of chars(passed to its process method) is an example of a sequence of a certain length repeated a certain number of times.
The class must have the following private instance variables:
● seqLength (final int) - holds the sought after length
● repeatNumber (final int) - holds the sought after length
● position (final int) - holds the start position in the DNA sequence (for tag reference)
● fail (boolean) - holds whether the processed sequence so far could not be this repeat type
● patternSoFar (String) - holds the accumulated chars so far given to this checker
● previous (QueueADT<Character>) - holds the initial chars that must later be checked against the later chars to determine if the sequence is repeated repeatNumber times
The class must have two constructors, implement the Checker interface, and override the toString method:
● public RepeatChecker(int seqLength, int repeatNumber)
o Initializes all instance variables: seqLength as specified, repeatNumber as specified, position as 0, fail as false, patternSoFar as “”, and previous as an empty queue
● private RepeatChecker(int seqLength, int repeatNumber, int position)
o Same as above but position is specified. This constructor is only used in the cloneHere method.
● public boolean process(char c)
o For the first seqLength of the characters, add them to the queue.
o After that see if they match the expected characters in the queue, if not, set the instance variable, fail, to true. (hint: after a character is matched dequeue and then enqueue)
o Return true if current length of the patternSoFar is the seqLength times repeatNumber and fail is false (meaning: it found the sought after pattern), otherwise return false.
● public boolean finished()
o Return fail
● public Checker cloneHere(int pos)
o Return a new RepeatChecker with pos
● public String toString()
o Return a String representation of this checker (see DnaTest.java for details)
o e.g. “Repeat(5,2) - 37{GCCATGCCAT}”
DnaAnalyser.java
This class uses the checkers to find patterns in DNA sequences. You will need to import java.util.Iterator at the top of this file.
The class must have the following private instance variables:
● checkers (LinkedQueue<Checker>) - holds all the checkers for this Analyser
● results (LinkedQueue<String>) - holds all the results for only the recently searched sequence
The class must have one constructor, and the following methods:
● public DnaAnalyser(LinkedQueue<Checker> checkers)
o Initializes all instance variables: checkers as specified, results as an empty LinkedQueue<String>
● public String search(Iterator<Character> dnaSequence)
o Returns a String of the entire dnaSequence searched
o fills the results queue with all of the patterns of the types in the checkers queue that were found.
o Below describes a detailed search algorithm
● public String printAnalysis(String dnaSequence)
o Prints out the results queue followed by a formatted dnaSequence string to visually confirm the results
Search Algorithm
Initialize a Queue for active checkers.
Clear previous DnaAnalysers’ results
Loop through each character from the dnaSequence (keeping track of its position)
Concatenate the character into a full String of the sequence
Add a new checker to the active checkers for each of the DnaAnalysers’ checkers using cloneHere(pos)
have all active checkers process(c)
If process(c) is successful add its toString() to the results
Otherwise if is not finished() leave this checker as an active checker
(HINT: Draw this algorithm with a few examples before implementing)
Marking Notes
Functional specifications
● Does the program behave according to specifications?
● Does it produce the correct output and pass all tests?
● Are the classes implemented properly?
● Does the code run properly on Gradescope (even if it runs on Eclipse, it is up to you to ensure it works on Gradescope to get the test marks)
Non-functional specifications
● Are there comments throughout the code (Javadocs or other comments)?
● Are the variables and methods given appropriate, meaningful names?
● Is the code clean and readable with proper indenting and white-space?
● Is the code consistent regarding formatting and naming conventions?
Penalties
● Lateness: 10% per day
● Submission error (i.e. missing files, too many files, etc.): 5%
● "package" line at the top of a file: 5%
● Compile or run-time error on Gradescope
● Failure to follow instructions, (i.e. changing variable types, etc.)
Remember you must do all the work on your own. Do not copy or even look at the work of another student. All submitted code will be run through cheating-detection software.
Submission (due Monday, December 6, 2021 at 11:55pm ET)
Rules
● Please only submit the files specified below.
● Do not attach other files even if they were part of the assignment.
● Do not upload the .class files! Penalties will be applied for this.
● Submit the assignment on time. Late submissions will receive a penalty of 10% per day.
● Forgetting to submit is not a valid excuse for submitting late.
● Submissions must be done through Gradescope and the test marks come directly from there. If your code runs on Eclipse but not on Gradescope, you will NOT get the marks! Make sure it works on Gradescope to get these marks.
● Assignment files are NOT to be emailed to the instructor(s) or TA(s). They will not be marked if sent by email.
● You may re-submit code if your previous submission was not complete or correct, however, re-submissions after the regular assignment deadline will receive a penalty.
Files to submit
● Checker.java
● PalindromeChecker.java
● RepeatChecker.java
● DnaAnalyser.java
Grading Criteria
Total Marks: [20]
Functional Specifications:
[2] PalindromeChecker.java
[2] RepeatChecker.java
[2] DnaAnalyser.java
[4] PalindromeChecker tests
[4] RepeatChecker tests
[4] DnaAnalyser tests
Non-Functional Specifications:
[0.5] Meaningful variable names, private instance variables
[0.5] Code readability and indentation
[1] Code comments
2021-12-08