CS-677 Assignment: Linear Models
Hello, dear friend, you can consult us at any time if you have any questions, add WeChat: daixieit
BU MET CS-677: Data Science With Python, v.2.0
CS-677 Assignment: Linear Models
Assignment
In this assignment, we will implement a number of linear mod- els (including linear regression) to model relationships between different clinical features for heart failure in patients.
For the dataset, we use ”heart failure clinical records data set at UCI: https://archive.ics.uci.edu/ml/datasets/Heart+failure+ clinical+records
Dataset Description: From the website: ”This dataset contains the medical records of 299 patients who had heart fail- ure, collected during their follow-up period, where each patient
profile has 13 clinical features.”
These 13 features are:
1. age: age of the patient (years)
2. anaemia: decrease of red blood cells or hemoglobin (boolean)
3. high blood pressure: if the patient has hypertension (boolean)
4. creatinine phosphokinase (CPK): level of the CPK enzyme in the blood (mcg/L)
5. diabetes: if the patient has diabetes (boolean)
6. ejection fraction: percentage of blood leaving the heart at each contraction (percentage)
7. platelets: platelets in the blood (kiloplatelets/mL)
8. sex: woman or man (binary)
9. serum creatinine: level of serum creatinine in the blood (mg/dL)
10. serum sodium: level of serum sodium in the blood (mEq/L)
11. smoking: if the patient smokes or not (boolean)
12. time: follow-up period (days)
target death event: if the patient deceased (DEATH EVENT = 1) during the follow-up period (boolean)
We will focus on the following subset of four features:
1. creatinine phosphokinase
2. serum creatinine
3. serum sodium
4. platelets
and try establish a relationship between some of them using various linear models and their variants.
Question 1:
1. load the data into Pandas dataframe. Extract two dataframes
with the above 4 features: df 0 for surviving patients (DEATH EVENT = 0) and df 1 for deceased patients (DEATH EVENT = 1)
2. for each dataset, construct the visual representations of correponding correlation matrices M0 (from df 0) and M1 (from df 1) and save the plots into two separate files
3. examine your correlation matrix plots visually and answer the following:
(a) which features have the highest correlation for surviving
patients?
(b) which features have the lowest correlation for surviving
patients?
(c) which features have the highest correlation for deceased patients?
(d) which features have the lowest correlation for deceased patients?
(e) are results the same for both cases?
Question 2: In this question you will compare a number of different models using linear systems (including linear regres- sion). You choose one feature X as independent variable X and another feature Y as dependent. Your choice of X and Y will depend on your facilitator group as follows:
1. Group 1: X: creatinine phosphokinase (CPK), Y : platelets
2. Group 2: X: platelets, Y : serum sodium
3. Group 3: X: serum sodium, Y : serum creatinine
4. Group 4: X: platelets, Y : serum creatinine
We will now look for the best model (from the list below) that best explains the relationship for surviving and deceased pa- tients. Consider surviving patients (DEATH EVENT = 0). Extract the corresponding columns for X and Y . For each of the models below, we will take 50/50 split, fit model with Xtrain and predict Ytest using Xtest . From the predicted val- ues Pred(yi ) we compute the residuals ri = yi - Pred(yi ). We can then estimate the loss function (SSE sum of the squared of residuals)
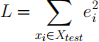
You do the same analysis for deceased patients. You will con- sider the following models for both deceased and surviving pa- tients:
1. y = ax + b (simple linear regression)
2. y = ax2 + bx + c (quadratic)
3. y = ax3 + bx2 + cx + d (cubic spline)
4. y = a log x + b (GLM - generalized linear model)
5. log y = a log x + b (GLM - generalized linear model)
For each of the model below, you will do the following (for both deceased and surviving patients)
(a) fit t he model on Xtrain
(b) print the weights (a, b, . . .)
(c) compute predicted values using Xtest
(d) plot (if possible) predicted and actual values in Xtest (e) compute (and print) the corresponding loss function
Question 3: Summarize your results from question 2 in a table like shown below:
|
Model |
SSE (death |
event=0) |
(death |
event=1) |
|
y = ax + b y = ax2 + bx + c y = ax3 + bx2 + cx + d y = a log x + b log y = a log x + b |
|
|
||
1. which model was the best (smallest SSE) for surviving pa- tients? for deceased patients?
2. which model was the worst (largest SSE) for surving pa- tients? for deceased patients?
2021-12-04
BU MET CS-677: Data Science With Python, v.2.0