Final Project Instructions
Hello, dear friend, you can consult us at any time if you have any questions, add WeChat: daixieit
Final Project Instructions
• Output for the project will be written in RMarkdown and submitted as a .rmd file
• Submit the .rmd file and any additional scripts in a .zip file
• Provide commentary on your code throughout your project
• Ensure your final project looks professional
• Projects are due December 14, 2023 at 5pm EST. Feel free to submit your project early!
Part 1: Background
Generalized linear models (GLM) is a term in statistics that describes a large family of regression models that are extensions of the ordinary linear regression model. GLMs characterize a linear relationship between response and predictor variables where the underlying distribution of the response variable is not normally distributed. In order to use GLMs appropriately, you must know the underlying distribution of the Y variable. We will explore the impact of model misspecification - that is, what happens if we use the wrong distribution.
We will simulation data from a Poisson distribution. The pdf of a Poisson distribution is given by:
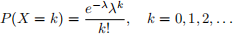
We will examine results from Poisson, Negative Binomial, and Normal regressions. Poisson regression, for example, uses the following formula:
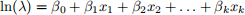
We can use our estimatedβ(ˆ)s to estimate the true Poisson parameter (which we set as part of our simulation), λ, and assess the bias of our model: Bias = λ(ˆ) - λ . We will do this for all three GLM models.
We will also consider the estimated variance of our β(ˆ)s. We will consider two different variance formulas:
• The naïve variance estimator: This estimator is our traditional variance estimate. The matrix formula
is Varnaïve(β(ˆ)) = (XTX)− 1 (XTX)− 1
• The robust variance estimator: This estimator is more robust to extreme values and heteroskedasticity. The matrix formula is: V arrobust(βˆ) = (XT X)−1XT ΩX(XT X)−1
The above formulas are so you can understand the mathematical underpinnings of this project, but we will use R functions to do all of this for us! Please do not hard code any complex formulas.
Part 2: Simulation Study
Create a simulation study to assess the impact of model misspecification on model bias and variance, including comparing the naïve and robust variances. Create a function to generate your simulation data and summarize your results (use multiple functions as needed). Carefully consider how to organize both your code scripts and your results.
Use the following conditions:
1. Sample sizes: N = 20, 100, 500
2. True Poisson parameter: λ = 4, 10
3. Run each simulation 10,000 times
You will also need to simulate your predictor variable. We will create a simple binary variable by simulating data from a binomial distribution where p=0.65. You only need to generate this data once.
x = rbinom (n, 1 , 0.65)
Using your simulated data, calculate your regression models: Poisson, normal, and negative binomial.
library(MASS) #need this package for the negative binomial regression
fit_pois = glm(y ~ x, family = poisson, data = dat)
fit_norm = glm(y ~ x, family = gaussian, data = dat)
fit_negb = glm.nb(y ~ x, data = dat)
Part2a: The β(ˆ)s
You will use your β(ˆ)s to estimate λ(ˆ) and then calculate the mean bias for each model. You can easily do to
this in R by using the predict function. You will need to set up your design matrix first:
#set up design matrix
new_data <- data.frame (intercept = 1 , x1 = x)
The code below will take your fitted models and return a λ(ˆ) for each model. You can then calculate the bias
of your λ(ˆ)s.
#calculate lambda hats
pois_lambda_hat = predict (fit_pois, newdata=new_data, type = "response") norm_lambda_hat = predict (fit_norm, newdata=new_data, type = "response") negb_lambda_hat = predict (fit_negb, newdata=new_data, type = "response")
Part2b: The Variances
You will need to obtain both variance estimators from your fitted models. The code below demonstrates how to obtain both variances from the Poisson model.
library (sandwich)
nvar_pois = vcov (fit_pois)[2 ,2]
rvar_pois = sandwich (fit_pois)[2 ,2]
Part 3: Results
Your summary of results should include:
1. A table summarizing your results on the bias of λ(ˆ) under the various scenarios
2. A table summarizing the naïve and robust variances under the various scenarios
3. A plot or plots showing the 95% confidence intervals for the beta coefficients using both the naïve and robust variances (i.e., two CIs per distribution). For simplicity, use the normal distribution for the CIs:
β(ˆ) ± 1.96 × SE(β(ˆ))
Recall you can obtain the SE(β(ˆ)) by taking the square root of the variance.
• The y-axis should be the β(ˆ)s, LBs, and UBs
• The x-axis should be the three probability distributions used (categorical x-axis)
• Facet on λ and N as needed
• Use color to improve the readability of your plot
• Provide a title, axis labels, etc. to make your plot informative
Part 4: Conclusions
Based on your simulation results, what conclusions can you draw? Consider questions like: What impact does misspecification have on the bias of ˆλ? How do the two variance estimators differ? How does sample size impact these results?
2023-12-11